科学家使用机器学习使抗药性检测更加有效
2020-04-21 17:48:00 来源:微生物研究所
细菌耐药性已成为影响全人类健康的一个主要问题,并引起了全世界的广泛关注。世界卫生组织提出的解决耐药性的措施之一是开发新的快速准确的耐药性诊断技术和相关试剂。传统的检测方法以细菌培养为基础,周期长,容易导致漏诊和误诊,延误最佳治疗时机。然而,基于基因的检测技术,如基因芯片和数字聚合酶链反应,灵敏、高效和快速,被认为是快速检测技术。然而,到目前为止,由于耐药基因型和表型的结果不一致,基因检测只能作为耐药检测培养方法的辅助手段。
中国科学院微生物研究所冯杰课题组等针对肺炎链球菌-内酰胺类耐药这一重要临床问题,采用机器学习方法挖掘耐药相关数据规律,建立基因型与表型的关系,使得基因检测不再是辅助手段,而是有望成为一种主要的快速耐药检测技术。
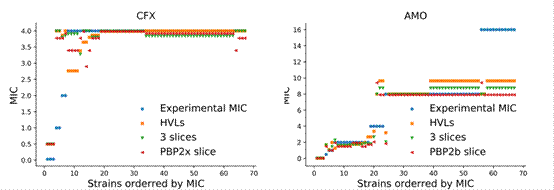
肺炎链球菌对-内酰胺类抗生素耐药的主要机制是三个pbp(PBP1a、PBP2b和PBP2x)的转肽酶结构域(TPD)的改变。由于来自不同临床肺炎链球菌分离株的多溴联苯醚的高可变性和链球菌之间重组引起的嵌合结构,多溴联苯醚极其多样,这使得难以将多溴联苯醚突变与临床耐药性联系起来。冯杰组的研究人员首先用类别方差法计算了NCBI数据库中公布的多溴联苯醚序列,获得了139个与耐药性高度相关的高变异氨基酸。以4300株肺炎链球菌的TPD序列和相应的头孢呋辛和阿莫西林耐药表型为数据库,其中80%作为训练集,20%作为测试集。HVLs用于预测头孢呋辛和阿莫西林的耐药水平。结果表明,预测效果与PBPs蛋白的TPD序列相当。进一步的分析表明,高密度脂蛋白与多溴联苯醚某些区域的序列有很强的相关性。因此,pbp2x的750 bp片段(2253 bp)和pbp2b的750 bp片段(2058 bp)可用于预测头孢呋辛和阿莫西林的耐药性。该长度仅需要一个桑格测序反应,这不仅使检测操作更简单,而且降低了成本。此外,通过检测人工构建的突变体和来自更多临床分离株的菌株的耐药性表型,进一步证实了机器学习方法能够准确预测耐药性表型。利用这种预测方法,研究人员对NCBI数据库中已测序的8138株肺炎链球菌进行了分析,建立了耐药表型、血清型和ST型之间的相关性,促进了对肺炎链球菌流行病学的了解。
该研究结果发表在《生物信息学简报》在线期刊上,中国南方科技大学的冯杰和杨亮教授是该通讯的共同作者。这项研究由国家自然科学基金和北京市科学技术委员会资助。